基因组编辑三大利器(生物通、生物探索)
来源:qnhu 时间:May 12, 2014, 8:42 p.m.http://www.ebiotrade.com/newsf/2013-10/2013101895100403.htm
http://www.biodiscover.com/news/research/109534.html
转录激活样效应因子核酸酶(transcription activator-like effector nuclease, TALEN)技术与锌指核酸酶(Zinc-finger nuclease, ZFN)技术组成了一大类强有力的基因组编辑工具,这一大类技术的发展重新划定了生物学研究的边界。这些嵌合核酸酶由两部分组成——一个可编码的序列特异性DNA结合模块与一个非特异性的DNA切割结构域。通过诱导DNA双链断裂(DNA double-strand break)来刺激容易出错的非同源末端连接或在特定基因所在的位置进行的同源定向修复,TALEN和ZFN能够完成一系列遗传学编辑修饰操作。
成簇规律间隔短回文重复(clustered regulatoryinterspaced short palindromic repeat, CRISPR)技术是最新出现的一种基因组编辑工具,它能够完成RNA导向的DNA识别及编辑。与其它基因组编辑工具相比,CRISPR技术更易于操作,具有更强的可扩展性。
本文将以上述三种技术为例,介绍并探讨新一代位点特异性基因组工程技术的生物学原理、未来发展趋势,及其在遗传学研究领域的作用和潜在的医学应用前景。
一、TALEN技术
TAL效应因子(TAL effector, TALE)最初是在一种名为黄单胞菌(Xanthomonas sp.)的植物病原体中作为一种细菌感染植物的侵袭策略而被发现的。这些TALE通过细菌 III类分泌系统(bacterial type III secretion system)被注入植物细胞中,通过靶定效应因子特异性的基因启动子来调节转录,来促进细菌的集落形成。由于TALE具有序列特异性结合能力,研究者通过将FokI核酸酶与一段人造TALE连接起来,形成了一类具有特异性基因组编辑功能的强大工具,即TALEN。
近年来, TALEN已广泛应用于酵母、动植物细胞等细胞水平基因组改造,以及拟南芥、果蝇、斑马鱼及小鼠等各类模式研究系统。2011年《自然•方法》(Nature Methods)将其列为年度技术,而2012年的《科学》(Science)则将TALEN技术列入了年度十大科技突破,针对该文的评论更是给予它基因组的巡航导弹技术的美誉。
1. TALEN结构及技术原理
1.1 TALEN的典型结构
如前文所述,典型的 TALEN由一个包含核定位信号(Nuclear localization signal, NLS)的N端结构域、一个包含可识别特定 DNA序列的典型串联TALE重复序列的中央结构域,以及一个具有FokI核酸内切酶功能的C端结构域组成。不同类型的TALEN元件识别的特异性DNA序列长度有很大区别。一般来说,天然的TALEN元件识别的特异性DNA序列长度一般为17-18bp;而人工TALEN元件识别的特异性DNA序列长度则一般为14-20bp。

图1 TALEN的结构。(A)与靶点DNA(灰色显示,PDB ID:3UGM)结合的TALE蛋白。每一个独立的TALE重复序列元件包含33到35个氨基酸残基,这些TALE重复序列元件能够通过两个高变异度的残基(即重复可变双残基,RVD,棍状显示)来识别一个单一的碱基对。(B)TALE核酸酶(TALEN)形成二聚体结合DNA的动画演示。TALEN目标位点由两个TALE结合位点组成,这两个位点间通过不同长度的间隔区序列(12-20bp)分开。 TALE可以被设计成仅仅识别左半侧位点或右半侧位点。图片来源: Thomas Gaj, Charles A. Gersbach, and Carlos F. Barbas III. (2013) ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 31(7): 397-405.
1.2 TALEN技术的原理与步骤
TALEN技术的原理并不复杂,即通过 DNA识别模块将TALEN元件靶向特异性的DNA位点并结合,然后在FokI核酸酶的作用 下完成特定位点的剪切,并借助于细胞内固有 的同源定向修复(HDR)或非同源末端连接 途径(NHEJ)修复过程完成特定序列的插入 (或倒置)、删失及基因融合(图2)。
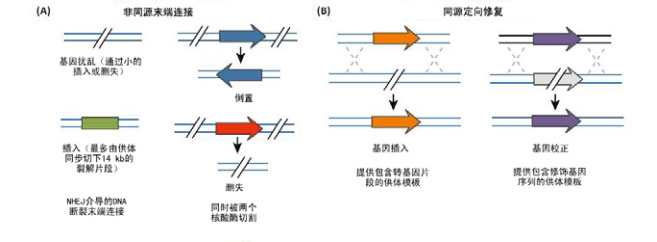
图2 TALEN进行基因组编辑的原理。利用位点特异性核酸酶可以进行基因组编辑,而核酸酶诱导的DNA双链断裂(DSB)可由同源定向修复(HDR)或非同源末端连接途径(NHEJ)来修复。(A)在供体质粒(donor plasmid)暴露出延长的同源臂(homology arm)的情况下,HDR可能导致插入的单个或多个转基因发生改变或取代原有的基因。(B)在缺失供体质粒的情况下,NHEJ介导的修复会产生小的插入或删失突变,并可能导致目标基因被破坏;在有双链寡核苷酸或线状供体质粒存在的情况下,这些DNA片段可能通过NHEJ介导的连接反应插入;同时诱导两个DSB的产生则会引起删失、插入和易位突变。图片来源:Thomas Gaj, Charles A. Gersbach, and Carlos F. Barbas III. (2013) ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 31(7): 397-405.
TALEN技术的核心原理就是在同一个蛋白(TALEN)上有序地实现引导进入细胞核、靶位点DNA的特异性识别和靶位点DNA的切割这三个不同的功能,这一点在上述TALEN典型结构一节中已作了较为详细的描述。在具体操作中,例如在实验室条件下,实现TALEN的关键就在于完成DNA的特异性识别功能,一般说来分为两个步骤。图3与图4分别以“铂金门”TALEN构建系统(Platinum Gate TALEN construction system)和商业化的easyT体系为例,展示了实验操作中TALEN元件的构建。
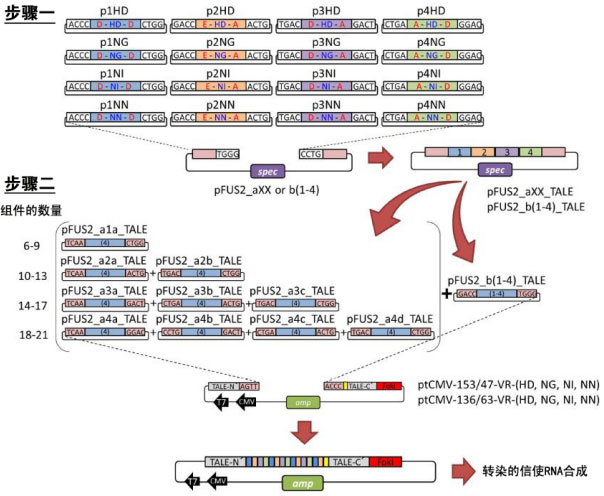
图 3 “铂金门”TALEN构建系统TALEN元件构建操作示意图。步骤一,四个或更少的组件被连接到阵列质粒(array plasmid)上;步骤二,构建好的阵列随后被连接到哺乳动物表达载体中;白色和粉色的长方形分别表示在BsaI和Esp3I限制性内切酶切割后留下的粘性末端;蓝色字母代表RVD,红色字母代表non-RVD变化,黄色长方形代表后一半重复。图片来源:Tetsushi Sakuma, Hiroshi Ochiai, Takehito Kaneko, Tomoji Mashimo, Daisuke Tokumasu, et al. (2014) Repeating pattern of non-RVD variations in DNA-binding modules enhances TALEN activity. Science Report, 3(3379): 1-8.
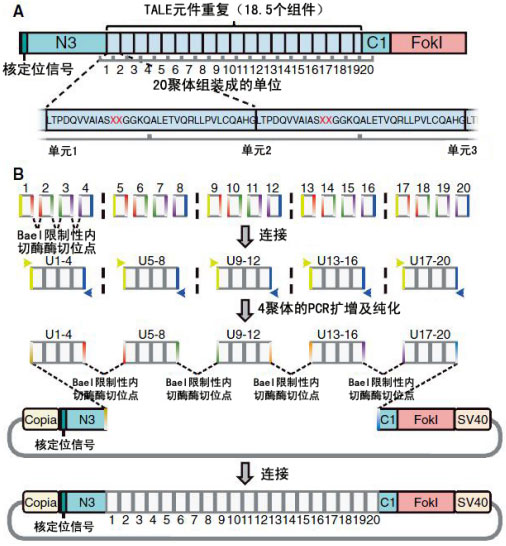
图 4 “easyT”TALEN构建系统TALEN元件构建操作示意图。(A)包含一个长度为18.5个组件的 TALE重复元件的TALEN体系示意图。该TALE重复元件由20个单体单位(monomer unit)组装而成。单体单位的边界在组装过程中发生了移位。(B)TALEN克隆示意图。第一步,由四个单体通过连接反应组装成4聚体;第二步,4聚体(4-mers)进行PCR扩增,琼脂糖凝胶电泳,胶回收并浓缩;最后,在第二次连接反应中,4聚体被组装到TALEN骨架质粒(backbone plasmid)上;黄色和蓝色箭头分别表示4聚体扩增时的正向引物与反向引物。图片来源:Tomonori Katsuyama, Arslan Akmammedov, Makiko Seimiya, Samuel C. Hess, Cem Sievers and Renato Par. (2013) An efficient strategy for TALEN-mediated genome engineering in Drosophila. Nucleic Acids Research, 41(17): e163-171.
1.2.1构建TAL靶点识别模块
TAL的DNA特异性识别单位是间隔32个恒定氨基酸残基的二联氨基酸。二联氨基酸与AGCT这4个核苷酸碱基有一一对应的关系:腺嘌呤(A)由NI识别、胸腺嘧啶(T)由NG识别、鸟嘌呤(G)由NN识别,而胞嘧啶(C)则由HD识别。实验操作中,我们通过 靶位点的DNA序列可以反推能特异性识别这一序列的二联氨基酸序列,从而构建TAL靶点识别模块。
1.2.2 TAL靶点识别模块的克隆与表达
根据之前对TALEN结构的介绍,我们需要将上一步骤中根据目标DNA序列构建好的一对TAL靶点识别模块与N端的核定位序列、C端的FokI酶连接起来,才能得到一个完整的TALEN元件。一般来说,我们可以采用专门用于构建TALEN的真核表达载体体系,将一对特异性的TAL靶点识别模块克隆进该载体中,再通过转染等方式导入细胞内。这种体系一般由供体质粒(donor plasmid,提供单基、二联及三联等类型的TAL模块)和骨架质粒(backbone plasmid,用于构建TALEN并表达构建好的TALEN)两类质粒构成,常用的TALEN体系有RCIscript-GoldyTALEN和pC-GoldyTALEN、TAL5-BB和pTAL6-BB及pCS2TAL3-DD和pCS2TALE-RR等。
2. TALEN技术的应用及近期发展
虽然 TALEN技术的基本原理并不难理解,但其发现过程却较为曲折。从1989年首次发现TAL起,研究者前后历时近21年才研究清楚TAL的工作原理。自2010年正式发明 TALEN技术以来,全球范围内多个研究小组利用体外培养细胞、酵母、拟南芥、水稻、果蝇及斑马鱼等多个动植物体系验证了TALEN的特异性切割活性。
2.1 TALEN技术的应用
2011年北京大学(Peking University)的 Zhang等人首次使用TALEN技术在斑马鱼中成功实现了定向突变和基因编辑;而爱荷华州立大学(Iowa State University)的Wang等人则在2012年,也以斑马鱼为模式动物,并首次使用TALEN技术在活体内完成了特定 DNA的删除、人工DNA插入等较为复杂的操作。随后TALEN技术在植物、大小鼠的基因组改造等方面的应用也顺利完成。而2013年 Zhang使用TALEN诱导了DNA双链断裂,提高同源定向修复效率,在斑马鱼中实现了同源重组基因打靶。
2.2 TALEN的近期发展
如前所述,经典的 TALEN体系已经广泛应用,越来越多的实验室以及实验外包公司均能很好地完成TALEN相关实验,但是基本限于单基因的插入或敲除操作,而且主要用于单个基因功能的研究。2013年,首尔国立大学化学系和国家基因工程创新举措研究中心的Kim课题组建立了一个全基因组规模(genome-scale collection)的TALEN体系,他们系统地选取了人类基因组中高度特异性的序列作为靶位点以避开脱靶(offtarget)效应,通过一种高通量克隆体系,一次性构建了18, 740个编码蛋白的基因的 TALEN质粒。
在这项研究中,研究者以一种巧妙的方式优化了TALEN质粒的结构,以检测插入靶位点后质粒对应位置上EGFP的表达的方式检测了TALEN靶位点插入成功率(图5a)。通过这一方式,他们可以研究不同间隔序列下特定靶位点插入效率(图 5b&c),从而针对每一个靶位点,都能选出最佳的TALEN体系结构。2014年2月,北京大学生命科学学院魏文胜课题组依托于一种自主研发的TALE蛋白组装技术(ULtiMATE system)完成了全部 TALE元件的解码工作。
近年来,随着TALEN技术逐渐成熟,全球范围内各实验室已广泛使用TALEN技术来 完成基因打靶操作。 TALEN通过与显微注跨越干细胞研究、基因治疗、神经网络,以及射、慢病毒感染等技术手段相结合,其应用范动植物育种等多个领域,强力推动生命科学的围越来越广。相信在不远的将来,其应用必将进步。
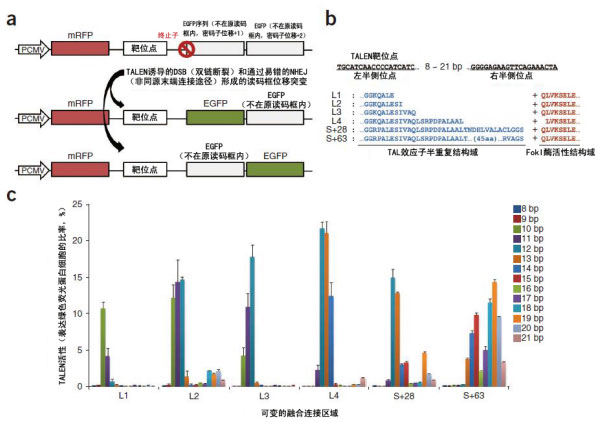
图 5 TALEN元件的优化。(a)基于RFP-GFP报告基因的各TALEN元件基因编辑活动的检测方法图示,只有插入靶位点后才能修正紧随其后EGFP的读码框以表达EGFP;(b)TALEN靶点和TAL效应子与FokI结构域融合连接区域的氨基酸序列;(c)TALEN基因编辑活力的比较。报告基因质粒包含靶位点,靶位点中有各类间隔序列(彩色标记),将报告基因质粒和TALEN质粒共转染到HEK293细胞中,然后使用流式细胞术分离GFP阳性的细胞。图片来源:Yongsub Kim, Jiyeon Kweon, Annie Kim, Jae Kyung Chon, Ji Yeon Yoo, Hye Joo Kim1, Sojung Kim et al. (2013) A library of TAL effector nucleases spanning the human genome. Nature Biotechnology, 31(3): 251-260.
二、ZFN技术
锌指核酸酶(Zinc-finger nuclease, ZFN)又名锌指蛋白核酸酶(ZFPN),它是一类人工合成的限制性内切酶,由锌指DNA结合域(zinc finger DNA-binding domain)与限制性内切酶的DNA切割域(DNA cleavage domain)融合而成。研究者可以通过加工改造ZFN的锌指DNA结合域,靶向定位于不同的DNA序列,从而使得ZFN可以结合复杂基因组中的目的序列,并由DNA切割域进行特异性切割。此外,通过将锌指核酸酶技术和胞内DNA修复机制结合起来,研究者还可以自如地在生物体内对基因组进行编辑。目前,在大量植物、果蝇、斑马鱼、蛙、大/小鼠及牛等物种中,ZFN技术已被广泛应用于靶向基因的突变,通过人工修改基因组信息可以产生遗传背景被修改的新物种。该技术在医学领域也具有非常重大的价值,对于疾病的基因治疗有潜在意义,具有非常广泛的应用前景。
1. ZFN结构及基本技术原理
1.1 ZFN的结构
顾名思义,ZFN由负责特异性识别序列的锌指DNA结合域和进行非特异性限制性内切酶切割的DNA切割域两部分组成(图6)。其中锌指DNA结合域部分一般包含3个独立的锌指(Zinc finger, ZF)重复结构,每个锌指结构能够识别3个碱基,因而一个锌指DNA结合域可以识别9bp长度的特异性序列(而ZFN二聚体,则包含6个锌指,可以识别18bp长度的特异性序列)。目前最常用的ZF结构为Cys2His2锌指,其结构由大约30个氨基酸包裹一个锌原子构成。研究表明,增加锌指的数量可以扩大ZFN特异性识别DNA序列的长度,从而获得更强的序列特异性。具体操作中,则一般通过模块化组合单个ZF,来获得特异性识别足够长的DNA序列的锌指DNA结合域。ZFN的三维空间结构如图6所示。
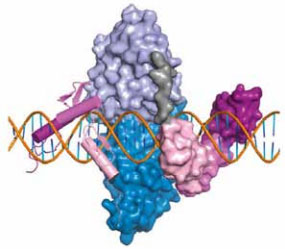
图6 ZFN的结构。该图为DNA双链与一对ZFN结合的示意图。每一对锌指用粉色标出,图像左侧的锌指用带状结构表示,右侧的锌指用填充空间结构表示;FokI的DNA切割域如蓝色区域所示;位于连接域与切割域之间的长度为四个氨基酸的“连接区”(linker)如灰色填充空间结构所示。DNA双链的糖-磷酸骨架为橙色,碱基显示为蓝色,在ZF结合位点两侧的DNA区域间距为6bp。该示意图由Smith等人在2000年根据锌指蛋白与DNA结合的晶体结构数据所编译而来。图片来源:Carroll D. (2011) Genome engineering with zinc-finger nucleases. Genetics. 188(4): 773-82.
此外,如果锌指DNA结合域与目的DNA序列能够完美配对,即便只含3个ZF结构的ZFN也能在基因组中特异性地结合18bp长度的序列。通过研究者长期的努力,识别每一种三联碱基的64种锌指组合中已有大部分被发现并编撰成目录,这些相关数据也都能够在公共的数据库或者文献中被检索到。针对每一条需要识别的目标序列,我们都可以使用与密码子对应的类似方式对锌指结构进行模块化组装(modular assembly),从而获得能够识别特定DNA序列的锌指蛋白结构。
ZFN的切割域与DNA结合域通过连接区(linker)结合。在ZFN中应用最广泛的DNA切割域来自IIS型限制性内切酶FokI。由于切割域与DNA链的结合能力较弱,因此DNA切割域必须以二聚体的形式发挥作用。构建锌指核酸酶时,应针对DNA各链上的邻近区域设计两条ZFN,使其DNA切割域能够位于双链的同一位置,以达到最佳的切割效果。两条ZFN之间具有被称为“间隔区”的spacer结构,该结构的长度以5~6bp为宜,7bp也能正常工作,合理的“间隔区”设计才能保证ZFN二聚体拥有最佳的工作空间(图7)。
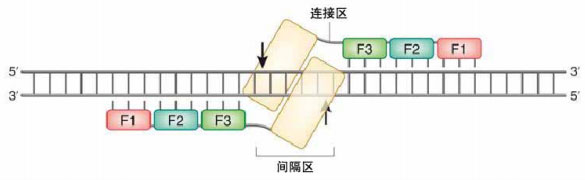
图7 ZFN特异性识别DNA并与DNA结合示意图。每个DNA识别域包含三个锌指,锌指从N端开始命名,在图中标示为F1、F2、F3。每个锌指结构分别与3个碱基发生直接接触,由此产生特异性。单独的FokI切割域不具有特异性识别能力,但当与锌指结构相连,并与另一个FokI切割域形成二聚体后,便能够对DNA双链进行切割。两个切割位点之间的距离约为4bp,如箭头所示。图片来源:Carroll D. (2011) Genome engineering with zinc-finger nucleases. Genetics. 188(4): 773-82.
1.2 ZFN技术的初步应用及其机制
ZFN技术可用于基因组编辑。针对目的基因序列设计并合成ZFN后,使之对DNA进行特异性切割,从而形成DNA双链断裂区(Double-StrandedBreaks,DSB);通过破坏非同源末端链接(non-homologous end joining, NHEJ)使目的基因失活,或借助同源重组(homologous recombination, HR)等方式完成DNA的修复连接,可以使断裂的DNA双链重新黏合。将以上两步结合起来便可以完成一般的基因组编辑操作,具体机制如图8所示。

图8 通过ZFN技术对基因进行编辑操作的几种主要方式示意图。通过对靶基因编码区域的目标序列进行ZFN切割,可以产生DSB,导致基因无法通过NHEJ修复而失活,从而达到基因敲除的目的。通过在两端引入wildtype(野生型,WT)DNA模板,并借助于同源重组(homologous recombination,HR)的修复,整个表达元件(expression cassette)可以替换发生DSB的区域,从而可以修复具有致病性的突变基因。通过提供能够覆盖ZFN切割位点附近整个核苷酸多态性区域的DNA序列,同样可以利用同源重组实现基因的修复,箭头显示的是ZFN切割位点。图片来源: Palpant NJ, Dudzinski D. (2013) Zinc finger nucleases: looking toward translation. Gene Ther, 20(2):121-7.
ZFN技术具有重大的应用价值。在科研和农业领域,该技术既可用于基因的敲除失活,也可用于导入目标基因,使基因激活或阻断,或者人为改造基因序列,使之符合人们的要求。在医疗领域,经ZFN技术改造后导入治疗性基因的质粒或干细胞可被导入人体,实现基因治疗。此外,ZFN技术也可以直接用于有害基因的修补替换或是直接删除,以达到相关治疗目的。ZFN技术具有极佳的特异性和效率,因此能将基因/基因组错误修改的风险降到最低。从理论上来说,研究人员甚至可以在任何物种中,对处于任意生长时期的细胞进行ZFN操作,可以自如地修改其基因,而还不破坏细胞状态。
2. ZFN技术的进展和应用
目前,除了传统的DNA重组技术外,合成具有可控特异性的锌指结构域的平台主要有两个:其一为Sangamo Bioscience公司所独有,在两个锌指的基础上生产更多 ZF的锌指核酸酶,并与Sigma公司合作,通过 Compozr项目销售预制的锌指;另一个是由锌指联盟(Zinc Finger Consortium)开发的 Oligomerized PoolENgineering(OPEN)技术,该技术主要通过大肠杆菌双杂交系统(E. coli two-hybrid selection system)来对ZFN进行筛选,这是一个开放平台,其模块化的锌指库和试剂皆可免费获得。
早期的 ZFN技术一般采用普通的 FokI-ZFN二聚体形式来保证其切割效率,但使用过程中可能因同源二聚体效应(homodimerization)而导致脱靶,影响切割特异性。Miller等人和Szczepek等人在2007年分别开发出FokI的变体,使得ZFN可以在异源二聚体(heterodimer)形式下使用,从而在保证切割效率的前提下大大提升了特异性,并减少了细胞毒性,具有更优良的科研和应用价值。
ZFN虽然是刚刚兴起的技术,但已被越来越广泛地应用到科研和医疗领域中。ZFN技术的先导之一,Sangamo Biosciences公司正在和宾夕法尼亚大学合作,研究ZFN技术通过介导核酸酶引起CCR5基因座的破坏。这一研究成果在治疗HIV中具有广阔的应用前景,而且ZFN技术也已尝试应用于杜氏肌营养不良症(Duchenne muscular dystrophy)、21三体综合征等遗传疾病的基因治疗(图9)。
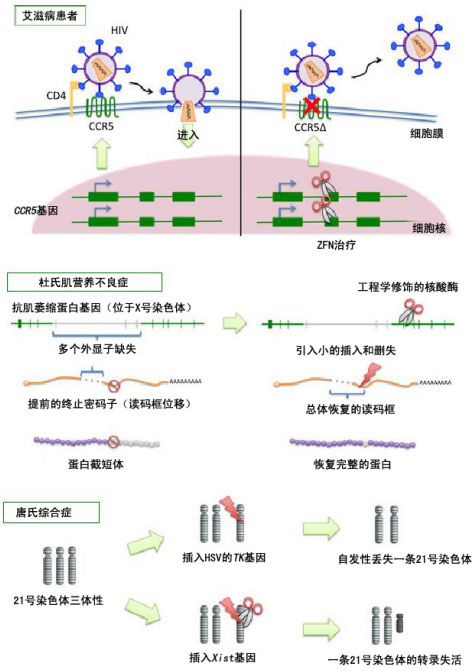
图9使用ZFN技术治疗艾滋病和遗传疾病的示意图。(上)治疗艾滋病:使用ZFN技术切割CCR5受体的编码序列,以破坏CCR5膜受体的功能,使HIV失去细胞感染能力。(中)治疗杜氏肌营养不良症:使用ZFN技术进行基因组编辑,插入小段序列将Dystrophin基因的读码框恢复正常。(下)治疗21三体综合征(唐氏综合症):使用ZFN插入HSV的TK基因,直接造成一条21号染色体自发丢失;使用ZFN在一条21号染色体上插入Xist基因,使这整个染色体失去功能。图片来源:Hongmei Lisa Li, Takao Nakano, and Akitsu Hotta. (2014) Genetic correction using engineered nucleases for gene therapy applications. Development Growth Differentiation, 56(1): 63-77.
早期的ZFN技术需要借助病毒或质粒载体的方式进入细胞,之后再表达形成具有功能的蛋白。但Barbas等人发现ZFN可以依靠自身锌指部分跨过细胞膜进入细胞,并发挥作用,如此则可避免载体插入重要基因而引起突变等潜在风险。近年来,一系列应用ZFN所取得的振奋人心的科研成果相继发表在高水平杂志上,如使患有人血液疾病乙型血友病(hemophilia B)的小鼠恢复血液凝结功能;在干细胞领域,研究者使用ZFN技术精确修正基因突变,从而使与人体疾病相关的缺陷蛋白失活等。
3.ZFN技术的缺陷
ZFN技术虽然简易实用,但也具有一定缺陷。ZFN对DNA的剪切需要两个FokI切割区域的二聚化,并且需要至少一个识别单元结合DNA。DNA识别域虽然具有较强的特异性识别能力,但由于ZFN剪切的过程并不完全依赖同源二聚体的形成,所以一旦形成异源二聚体,就很可能造成脱靶效应,并最终可能导致DNA的错配和序列改变,产生较强的细胞毒性。当这些不良影响积累过多,超过细胞修复机制承受的范围时,便会引起细胞的凋亡。另一方面,该手段仍然受到现有生物学领域研究手段的限制,因此在细胞内部操作的精确程度和后果都较难预料。如果ZFN引起相关基因突变,则可能会导致一系列意想不到的后果,在与人体相关的应用领域,甚至可能引发癌症。另外,ZFN作为基因治疗的手段之一,如果在生物体内使用,可能会引发免疫反应。现有的研究手段尚不能预测引入的ZNF蛋白是否会引起免疫系统的进攻。并且到目前为止,ZFN技术只能用于体外操作(in vitro),在对人体提取的细胞进行处理之后,再导入回输到病人体内。而直接向患者体内导入相关ZFN元件进行基因编辑处理则具有较大的潜在风险,且效率不高。以上诸多限制导致人体相关的ZFN操作较为繁琐,难以推广应用。
三、CRISPR/Cas系统
不论是TALEN技术还是ZFN技术,其定向打靶都依赖于DNA序列特异性结合蛋白模块的合成,这一步骤非常繁琐费时。而CRISPR/Cas技术作为一种最新涌现的基因组编辑工具,能够完成RNA导向的DNA识别及编辑。CRISPR/Cas技术使用一段序列特异性向导RNA分子(sequence-specific guide RNA)引导核酸内切酶到靶点处,从而完成基因组的编辑。CRISPR/Cas系统的开发为构建更高效的基因定点修饰技术提供了全新的平台。
1.CRISPR/Cas系统元件与特征
CRISPR/Cas系统最早是在细菌的天然免疫系统内发现的,其主要功能是对抗入侵的病毒及外源DNA。1987年大阪大学(OsakaUniversity)的研究人员在E.coliK12的碱性磷酸酶基因附近发现了成簇的规律间隔的短回文重复序列(Clustered regularly interspaced short palindromic repeat, CRISPR),其结构如图10所示,目前普遍认为有40%的细菌基因组具有这样的结构。

图10 CRISPR的结构(以嗜热链球菌LMD-9基因组CRISPR1/Cas系统的位点为例)。(上)Cas基因由蓝色表示,包括广泛存在的cas1和cas2,II类系统特征基因cas9和csn2。重复间隔物阵列(CRISPR)由黑色表示。(下)CRISPR的重复序列(repeat)和间隔物序列(spacer)分别用黑色菱形、灰色长方形表示。缩写:L,前端;T,末端重复;数字代表间隔物序列被获取的顺序。图片来源:Rodolphe Barrangou1 and Philippe Horvath. (2012) CRISPR: New Horizons in Phage Resistance and Strain Identification. Annual Review of Food Science, 3: 143-162.
CRISPR/Cas系统由CRISPR序列元件与Cas基因家族组成。其中CRISPR由一系列高度保守的重复序列(repeat)与同样高度保守的间隔序列(spacer)相间排列组成。而在CRISPR附近区域还存在着一部分高度保守的CRISPR相关基因(CRISPR-associated gene, Cas gene),这些基因编码的蛋白具有核酸酶活性的功能域,可以对DNA序列进行特异性的切割。
2. CRISPR/Cas系统工作原理
CRISPR/Cas作为原核生物中普遍存在的一种系统,最初的功能就是识别外源性入侵的核酸序列,并对其进行特异性降解,以达到抗病毒的作用。这一过程分两步进行——crRNA的合成及在crRNA引导下的RNA结合与剪切,具体机制如图11所示,包含crRNA的生物学合成和RNA的结合与剪切两大步骤。
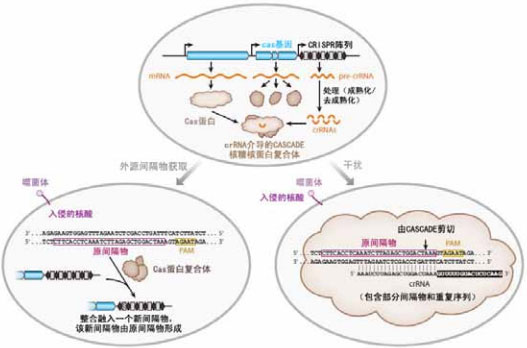
图11 CRISPR抗病毒运行机制。(上)crRNA和Cas蛋白的生物学合成:Cas基因转录为mRNA,随后翻译为Cas蛋白,Cas蛋白可以形成CASCADE复合体(抗病毒防御的CRISPR相关复合体)。CRISPR重复间隔物阵列转录为全长的前体crRNA(pre-crRNA),随后经过加工成为crRNA。这些crRNA包含部分的重复间隔序列。(下左)间隔物(spacer)获取:噬菌体的原间隔物序列,一般在PAM(原间隔物模块)的旁边,可由Cas蛋白识别,并产生一个新的重复间隔物单元,插在原有的重复间隔物阵列前端。(下右)干扰:由crRNA介导的CASCADE核糖核蛋白复合体识别入侵的同源序列,在PAM附近的原间隔物序列处将这些双链DNA(dsDNA)截断。图片来源:Rodolphe Barrangou1 and Philippe Horvath. (2012) CRISPR: New Horizons in Phage Resistance and Strain Identification. Annual Review of Food Science, 3: 143-162.
2.1 crRNA的生物学合成
CRISPR区域第一个重复序列上游有一段CRISPR的前导序列(Leader sequence),该序列作为启动子来启动后续CRISPR序列的转录,转录生成的RNA被命名为CRISPRRNA(简称crRNA)。
2.2 RNA的结合与剪切
CRISPR/Cas系统中crRNA与tracrRNA(反式激活的crRNA)形成嵌合RNA分子,即单向导RNA(Single guide RNA,sgRNA)。sgRNA可以介导Cas9蛋白在特定序列处进行切割,形成DNA双链断裂(Double-Stranded Break, DSB),完成基因定向编辑等的各类操作。
3. 不同类型的CRISPR/Cas系统
根据功能元件的不同,CRISPR/Cas系统可以分为I类系统、II类系统和III类系统。这三类系统又可以根据其编码Cas蛋白的基因不同而分为更多的亚类。不同类型CRISPR/Cas系统完成干扰的步骤也有所不同(图12)。
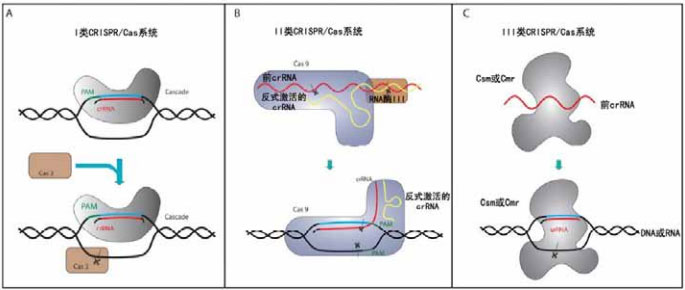
图12 三种不同的CRISPR/Cas干扰系统作用步骤。CRISPR/Cas系统根据分类有三种,其共同特点是都具有DNA区域(蓝色)、靶向crRNA(红色)和原间隔物模块(PAM,绿色)。在I类系统(A)中,入侵的DNA有Cascade:crRNA复合体识别,PAM模块则能促进外源性DNA的识别,随后核酸酶Cas3被募集并将目标DNA降解。在II类系统(B)中,只需要单独的Cas9蛋白即可完成干扰,并不依赖一个多蛋白复合体,Cas9和反式激活的crRNA(tracrRNA)、前crRNA(pre-crRNA)形成复合体,该复合体促使RNA酶III将前crRNA加工为成熟的crRNA。在III类系统(C)中,一个多蛋白复合体(Csm或Cmr)或Cas6促进前crRNA转化为成熟的crRNA,最终导致目标DNA的降解。图片来源:Hagen Richter, Lennart Randau and André Plagens. (2013) Exploiting CRISPR/Cas: Interference Mechanisms and Applications. International Journal of Molecular Science, 2013, 14, 14518-14531.
I类和III类CRISPR/Cas系统进行干扰时只需要crRNA和Cas蛋白两种元件的参与,而II类CRISPR/Cas系统包括crRNA、tracrRNA和Cas蛋白三种元件。其中II类CRISPR/Cas系统最先在改造后用于小鼠和人类基因组编辑,同时也是目前研究最为充分的系统。根据Cas蛋白的类型不同分为三个亚类:II-A类含有Cas1、Cas2、Cas9和Csn2样蛋白;II-B类含有Cas1、Cas2、Cas4和Csx12样Cas9四种蛋白;II-C类则有Cas1、Cas2及Cas9三种蛋白。此外,II类CRISPR/Cas系统也是目前最常用于人工基因组编辑的CRISPR/Cas系统,其靶向基因组编辑的步骤如图13所示。

图13 利用一段小向导RNA(sgRNA):Cas复合体系统靶向基因组编辑的步骤。将编码密码子优化的Cas9(红色)序列、一段核定位序列(NLS)和一段包括目标靶序列的小向导RNA(sgRNA,黄色)序列同时构建在一个质粒中,再将质粒转染进目标细胞。一个有功能的sgRNA:Cas9干扰复合体会在细胞内完成组装,该复合体会在PAM结构的上游目标DNA序列上诱导产生一个双链断裂(DSB),而DSB则能被宿主细胞的DNA修复系统、同源重组系统(HR)和非同源末端连接途径(NHEJ)修复。HR系统以宿主的等位基因为模板复原野生型序列,将序列恢复为断裂前的状态;而容易出错的NHEJ系统则会在目标位点(灰色)引入插入和删失。使用一段合成的供体DNA模板与Cas系统质粒共转染,可以诱导HR(蓝色),提高编辑效率。图片来源:Hagen Richter, Lennart Randau and André Plagens. (2013) Exploiting CRISPR/Cas: Interference Mechanisms and Applications. International Journal of Molecular Science, 2013, 14, 14518-14531.
4. CRISPR技术的应用
自1987年大阪大学(Osaka University)的研究人员在细菌的天然免疫系统中发现CRISPR/Cas系统以来,CRISPR作为一种潜在技术在很长时间内都没有得到重视与发展。近年来,由于基因工程技术的突飞猛进,CRISPR/Cas俨然已经成为科学界最炙手可热的热点之一,被广泛应用于各类体内和体外体系的遗传学改造、转基因模式动物的构建,甚至基因治疗领域。
2013年初的《科学》第339卷第6121期连载了两篇具有重要意义的CRISPR技术论文,其中一篇描述的是麻省理工学院(Massachusetts Institute of Technology, MIT)Zhang的研究组使用CRISPR技术完成了多重基因组编辑,另一篇描述了哈佛医学院(Harvard Medical School)Church的研究组首次使用CRISPR技术完成了RNA介导的人类基因组编辑(图14)。他们使用基因工程学方式修改了细菌的II类CRISPR系统,并比较了这种新系统与传统TALEN方法在基因组编辑方面的效率差异,结果发现这种方式比TALEN有更快的时效性。同时,该研究组还建立了一个覆盖约40.5%外显子的基因组水平的gRNA群。

图14 使用一种基因工程修饰的II类CRISPR系统完成了人类细胞的基因组编辑。(A)人类细胞的RNA介导基因打靶涉及C末端包含SV40核定位信号(nuclear localization signal)的Cas9蛋白和一个或一个以上的向导RNA(guide RNA, gRNA)的共表达(上半部分共表达两个质粒的结构示意图),这一过程由人类U6聚合酶III(U6 polymerase III)的启动子介导。Cas完成DNA双链的解聚并在gRNA(guide RNA)的识别下切割特定的DNA双链位置,该过程前提是其3’端有序列正确的原间隔物模块(protospacer-adjacent motif, PAM)。原则上任何符合GN20GG序列模式的基因组序列都可以被特异性靶向识别(下半部分靶向识别的作用机制示意图)。(B)一个基因组整合的GFP编码序列被一个终止密码子和一个长达68bp的基因组片段在AAVS1位点的插入打断,使用合适的供体序列通过同源重组(HR)的方式修复GFP序列能诱使GFP功能恢复,形成GFP阳性的细胞,之后则可通过流式细胞术(FACS)分离。T1和T2向导RNA靶向序列定位于AAVS1片段区域。TALEN元件的两个单体的结合位点用上划线表示。图片来源:DiCarlo, Julie E. Norville1, George M. Church. (2013) RNA-guided human genome engineering via Cas9. Science, 339(6121): 823-826.
同年,中国科学院动物研究所周琪研究员利用CRISPR-Cas技术在大鼠中实现了多基因同步敲除;而怀特海德研究所(Whitehead Insititute)的 Jaenisch利用CRISPR-Cas技术构建了条件敲除的小鼠转基因模型;北京大学生命科学学院的瞿礼嘉教授课题组利用CRISPR-Cas系统成功地实现了对水稻特定基因的定点突变;杜克大学Pratt工程学院基因组科学研究所的Gersbach研究组则已经开始尝试使用CRISPR技术进行基因治疗。仅这一年内,CRISPR/Cas领域就取得了如此多鼓舞人心的突破,简直可以用一日千里来形容了。
四、三种基因定点修饰技术的总结与比较
1. TALEN、ZFN和CRISPR/Cas的共同点
从分子生物学角度看来,基因定点修饰操作可以分为敲入(knock in)、敲除(knock out)、删失(deletion)及基因融合(gene integration)这几种类型。而其中敲除又有多重敲除(multiplex knockout)和条件敲除(conditional knockout)等特殊类型,本质上均是利用非同源末端链接途径(NHEJ)修复和同源重组(HR)修复,联合特异性DNA的靶向识别及核酸内切酶完成的DNA序列改变,其总体模式如图15所示。
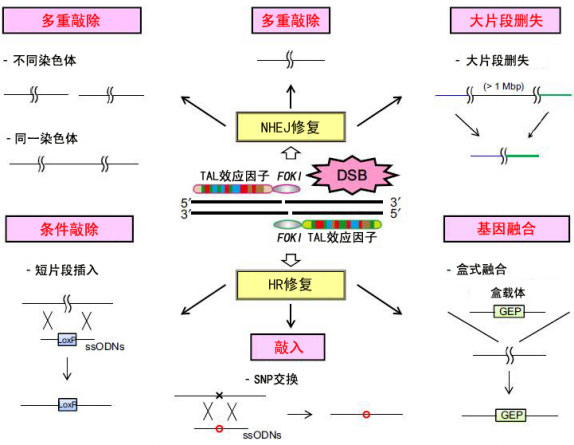
图15 使用 TALEN、ZFN和CRISPR等技术的分子生物学途径示意图。图片来源:Tomoji Mashimo. (2014) Gene targeting technologies in rats: Zinc finger nucleases, transcription activator-like effector nucleases, and clustered regularly interspaced short palindromic repeats. Development Growth Differentiation, 56(1): 46–52.
近年来TALEN、ZFN和CRISPR/Cas三大基因定点修饰技术已经广泛应用于生命科学与医学的各个方面,包括但不局限于转基因动植物模型的构建、基因治疗及转基因育种等。虽然TALEN、ZFN和CRISPR/Cas三种技术在技术细节上有着各自独一无二的特色,但它们在各类应用中的基本模式却是相似的,例如在转基因大鼠的构建上,三种技术均是以显微注射的方式进入大鼠胚胎的(图16)。
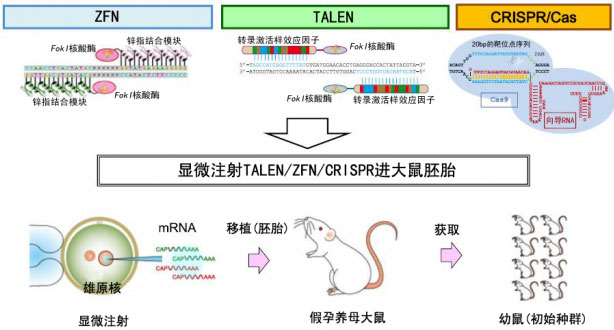
图16 使用基因工程学方式构建基因靶向敲除大鼠的示意图。在大鼠(rat)胚胎中以显微注射的方式使用TALEN、ZFN和CRISPR等技术进行基因打靶。图片来源:Tomoji Mashimo. (2014) Gene targeting technologies in rats: Zinc finger nucleases, transcription activator-like effector nucleases, and clustered regularly interspaced short palindromic repeats. Development Growth Differentiation, 56(1): 46–52.
2. TALEN、ZFN和CRISPR/Cas的技术特点
虽然TALEN、ZFN和CRISPR/Cas均能用于与基因组定点修饰相关的各类操作,应用范围有很大程度的重合,但是这三种技术有各自不同的技术特点和适用范围(表1),因此实际操作中,实验者都会根据实际需要选择合适的基因组定点修饰技术方案。
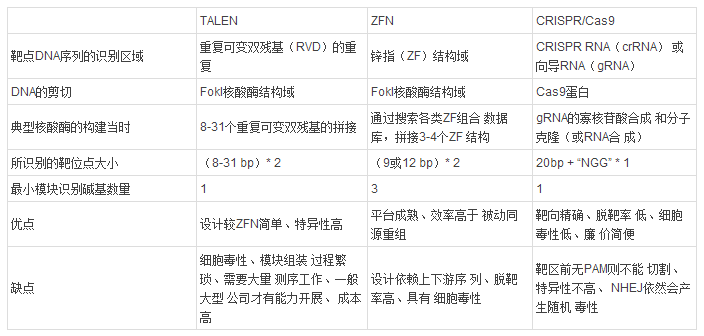
表1 TALEN、ZFN和CRISPR/Cas三种基因定点修饰技术特点的比较,表格来源:Hongmei Lisa Li, Takao Nakano, and Akitsu Hotta. (2014) Genetic correction using engineered nucleases for gene therapy applications. Development Growth Differentiation, 56(1): 63-77.
TALEN技术是目前商业化最成功的技术,虽然将单个的TALEN模块进行组装需要大量的分子克隆和测序操作,十分繁琐,但是很多商业公司可以提供组装好的三联密码子TALEN模块,甚至四联密码子TALEN模块,这样就大大缩短了构建TALEN元件的实验周期。不过也正是因为如此,绝大多数实验室都难以自行完成TALEN技术的完整操作,对其推广造成了障碍。
ZFN技术则是最早被广泛使用的基因组定点修饰技术,各大平台均比较完善,有很多可以直接使用的资源,然而由于其自身的三联属性,其设计比TALEN更为繁琐,而且高度依赖于目标序列及其上下游序列,还具有脱靶率高及细胞毒性大等诸多限制性因素。
CRISPR/Cas技术摆脱了合成并组装具有特异性DNA识别能力蛋白模块的繁琐操作,其gRNA的设计和合成工作量远远小于TALEN和ZFN技术的DNA识别模块的构建过程,且毒性远远低于ZFN技术。然而CRISPR/Cas技术也有上下文依赖性,目前只能应用于上游有PAM序列的靶位。
TALEN、ZFN和CRISPR/Cas三大基因组定点修饰技术应用于各个生物医学领域的历史都并不长,但近年来发展无比迅速,积累了大量的网络资源和平台资源。以模式动物果蝇(Drosophila melanogaster)的基因组定点修饰为例,目前可查的基因组定点修饰相关数据库已经多达十七个(表2)。
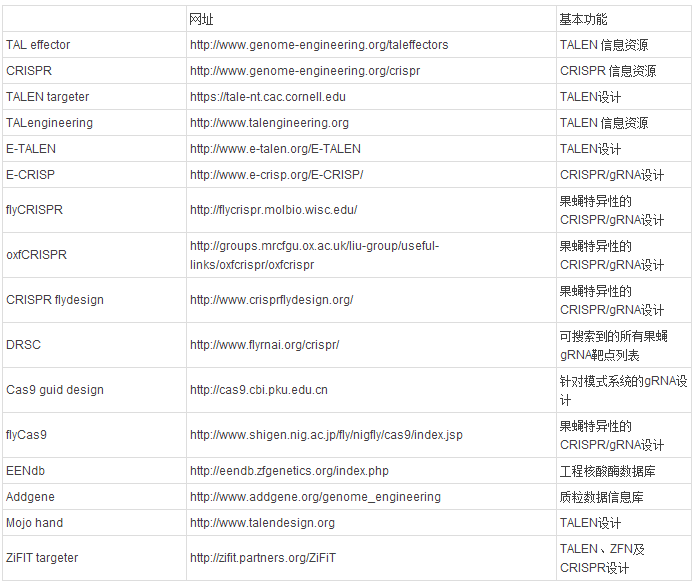
表2 现有的TALEN、ZFN及CRISPR相关设计数据库资源,表格来源:Kelly J. Beumer, and Dana Carroll. (2014) Targeted genome engineering techniques in Drosophila. Methods, in press, DOI: 10.1016/j.ymeth.2013.12.002.
由于在技术特征方面存在区别,TALEN、ZFN和CRISPR/Cas作为不同的技术在研究领域上虽然有极高重复度,但一些特殊的研究领域,在这几种基因组修饰工具的选择上,依然具有较强的偏好性。例如在基因治疗领域,不同技术在应用上也有不同的分工(表3)。
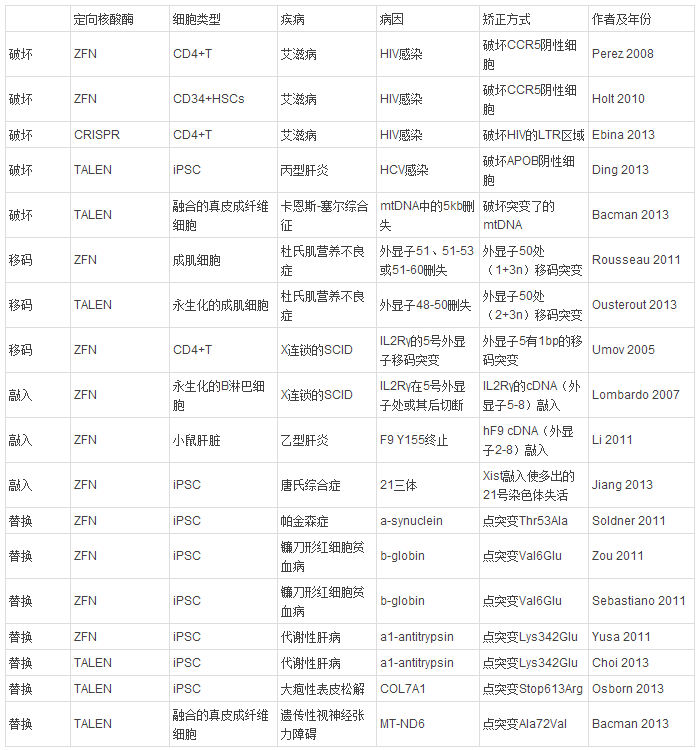
表3 工程修饰的核酸酶在人类遗传病治疗中的研究
参考文献
1. Thomas Gaj, Charles A. Gersbach, and Carlos F. Barbas III. (2013) ZFN, TALEN, and CRISPR/Cas-based methods for genome engineering. Trends in Biotechnology, 31(7): 397-405.
2. Yongsub Kim, Jiyeon Kweon, Annie Kim, Jae Kyung Chon, Ji Yeon Yoo, Hye Joo Kim, Sojung Kim, Choongil Lee, et al. (2013). A library of TAL effector nucleases spanning the human genome. Nature Biotechnology, 31(3): 233-239.
3. Yongsub Kim, Jiyeon Kweon, Annie Kim, Jae Kyung Chon, Ji Yeon Yoo, Hye Joo Kim1, Sojung Kim, et al. (2013) A library of TAL effector nucleases spanning the human genome. Nature Biotechnology, 31(3): 251-260.
4. Chuanxian Wei, Jiyong Liu, Zhongsheng Yu, Bo Zhang, Guanjun Gao, Renjie Jiao. (2013) TALEN or Cas9 e Rapid, Efficient and Specific Choices for Genome Modifications. Journal of Genetics and Genomics, 40(1): 281-289.
5. Qiurong Ding, Youn-Kyoung Lee, Esperance A.K. Schaefer, Derek T. Peters1, Adrian Veres, Kevin Kim, Nicolas Kuperwasser, et al. (2012) A TALEN genome-editing system for generating human stem cell-based disease models. Cell Stem Cell, 12(2): 238–251.
6. Victoria M. Bedell, Ying Wang, Jarryd M. Campbell, Tanya L. Poshusta, Colby G. Starker, Randall G. Krug II, et al. (2012) In vivo genome editing using a high-efficiency TALEN system. Nature, 491(7422): 114-118.
7. Yang J, Zhang Y, Yuan P, et al. Complete decoding of TAL effectors for DNA recognition. Cell research, 2014, in press.
8. Dana Carroll. (2011) Genome engineering with zinc-finger nucleases. Genetics, 188(4): 773-82.
9. Dana Carroll. (2008) Zinc-finger Nucleases as Gene Therapy Agents. Gene Therapy, 15(22): 1463–1468.
10. Palpant NJ, Dudzinski D. (2013) Zinc finger nucleases: looking toward translation. Gene Therapy, 20(2):121-7.
11. Severine Remy, Laurent Tesson, Severine Menoret, Claire Usal, Andrew M. Scharenberg, Ignacio Anegon. (2010) Zinc-finger nucleases: a powerful tool for genetic engineering of animals. Transgenic Research, 19(1): 363–371.
12. Eva-Maria Händel, and Toni Cathomen. (2011) Zinc-Finger Nuclease Based Genome Surgery: It’s All About Specificity. Current Gene Therapy, 11(1): 28-37.
13. NJ Palpant, and D Dudzinski. (2013 ) Zinc finger nucleases: looking toward translation. Gene Therapy, 20: 121-127.
14. Seokjoong Kim, and Jin-Soo Kim. (2011) Targeted genome engineering via zinc finger nucleases. Plant Biotechnology Reports, 5(1): 9-17.
15. Wu J, Kandavelou K, and Chandrasegaran S. (2007) Custom-designed zinc finger nucleases: what is next? Cellular and Molecular Life Sciences, 64(22): 2933-2944.
16. Rodolphe Barrangou1, and Philippe Horvath. (2012) CRISPR: New Horizons in Phage Resistance and Strain Identification. Annual Review of Food Science, 3(1): 143-162.
17. Hagen Richter, Lennart Randau, and André Plagens. (2013) Exploiting CRISPR/Cas: Interference Mechanisms and Applications. International Journal of Molecular Science, 14(1): 14518-14531.
18. Le Cong, Ann Ran, David Cox, Shuailiang Lin, Robert Barretto, Naomi Habib, et al. (2013) Multiplex genome engineering using CRISPR/Cas systems. Science, 339(6121): 819-823.
19. Prashant Mali1, Luhan Yang, Kevin M. Esvelt, John Aach, Marc Guell1, James E. DiCarlo, Julie E. Norville1, George M. Church. (2013) RNA-guided human genome engineering via Cas9. Science, 339(6121): 823-826.
20. Wenyan Jiang, David Bikard, David Cox, Feng Zhang, and Luciano A Marraffini. (2013) RNA-guided editing of bacterial genomes using CRISPR-Cas systems. Nature Biotechnology, 31(3): 233-239.
21. Woong Y Hwang, Yanfang Fu, Deepak Reyon, Morgan L Maeder, Shengdar Q Tsai, Jeffry D Sander, Randall T Peterson, J-R Joanna Yeh, and J Keith Joung. (2013) Efficient genome editing in zebrafish using a CRISPR-Cas system. Nature Biotechnology, 31(3): 227-229.
22. Judith Reeks, James H. Naismith, and Malcolm F. White. (2013) CRISPR interference: a structural perspective. Biochemistry Journal, 453(1): 155–166.
23. Kelly J. Beumer, Jonathan K. Trautman, Michelle Christian, Timothy J. Dahlem, Cathleen M. Lake, R. Scott Hawley, David J. Grunwald, Daniel F. Voytas, and Fand Dana Carroll. (2013) Comparing zinc finger nucleases and transcription activator-like effector nucleases for gene targeting in Drosophila. G3: Genes Genomes Genetics, 3(10): 1717-1725.
24. Hongmei Lisa Li, Takao Nakano, and Akitsu Hotta. (2014) Genetic correction using engineered nucleases for gene therapy applications. Development Growth Differentiation, 56(1): 63-77.
25. Tomoji Mashimo. (2014) Gene targeting technologies in rats: Zinc finger nucleases, transcription activator-like effector nucleases, and clustered regularly interspaced short palindromic repeats. Development Growth Differentiation, 56(1): 46–52.
26. Kelly J. Beumer, and Dana Carroll. (2014) Targeted genome engineering techniques in Drosophila. Methods, in press, DOI:10.1016/j.ymeth.2013.12.002.
27. Tomonori Katsuyama, Arslan Akmammedov, Makiko Seimiya, Samuel C. Hess, Cem Sievers and Renato Par. (2013) An efficient strategy for TALEN-mediated genome engineering in Drosophila. Nucleic Acids Research, 41(17): e163-171.
28. Tetsushi Sakuma, Hiroshi Ochiai, Takehito Kaneko, Tomoji Mashimo, Daisuke Tokumasu, et al. (2014) Repeating pattern of nonRVD variations in DNA-binding modules enhances TALEN activity. Science Report, 3(3379): 1-8.
下一篇:The dawn of evolutionary genome engineering
